L’équipe ‘Sciences Analytiques des Biomolécules’ de l’IBMM (F12), dirigée par C. Enjalbal, fait partie du Département « Acides Aminés, Peptides & Protéines » (DAPP).
L’équipe regroupe actuellement 11 permanents (6 CE, 1C, 3 BIATSS et 1 ITA) dont les expertises sont toutes liées aux sciences analytiques.
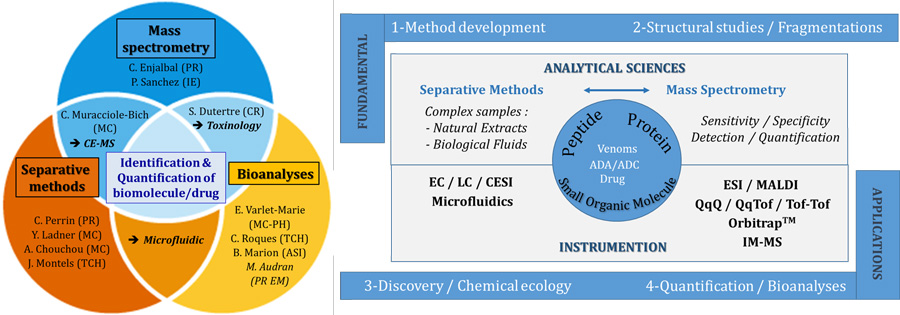
L’équipe F12 bénéficie d’expertises complémentaires pour aborder des problématiques analytiques en lien avec l’écologie chimique et en la pharmacie. De nombreuses études de recherches fondamentales et applicatives utilisant la spectrométrie de masse, la chromatographie liquide, l’électrophorèse capillaire et la microfluidique sont menées pour la détection et la quantification dans des échantillons complexes de peptides, de protéines, de toxines, d’anticorps, ainsi que de petits médicaments organiques, dans le cadre de problématiques pharmaceutiques et thérapeutiques (études de fragmentation, optimisation/validation de méthodes de détection et de quantification).
Afin de refléter la diversité/complémentarité des expertises scientifiques présentes au sein de l’équipe F12, l’équipe est organisée en trois groupes : Spectrométrie de masse, Méthodes séparatives et Stratégies omiques.
L’équipe dispose au sein de ses laboratoires d’un parc analytique conséquent comprenant des instruments séparatifs (1 HPLC, 5 CE, 1 CESI) pouvant être associés à plusieurs spectromètres de masse situés de la plateforme LMP. Ces instruments sont équipés d’une source d’ionisation ambiante (ESI/APCI) et présentent différentes configurations d’analyseurs de masse (piège à ions (IT) pour les dissociations itératives (ESI-MSn), triple quadripôle (QqQ) pour les bioanalyses (ESI-MS/MS), temps de vol (QTof) couplé à la mobilité ionique pour les analyses multimodales à haute résolution (ESI-IM-MS/MS)).
L’équipe F12 bénéficie d’expertises complémentaires lui permettant de mener une recherche fondamentale et applicative liée aux questions pharmaceutiques et thérapeutiques impliquant des analyses de peptides et de protéines en tant que biomolécules ciblées, et dans une moindre mesure de petits composés organiques (médicaments, substances dopantes) en tant que composés bioactifs. Ainsi, la plupart des sujets de recherche de F12 sont liés au développement de méthodes pour des analyses qualitatives et quantitatives dans des milieux biologiques complexes (plasma, urine, lysats cellulaires, extraits naturels tels que les venins).

Nos Thématiques
Sonder la chiralité dans les peptides thérapeutiques
Sonder la chiralité dans les peptides thérapeutiques : Effect of Asp, His and Gly ? – GLYMS (AAPG 2023 ANR – 2023 2026). Porteur
Le projet GLYMS vise à fournir des outils analytiques pour aborder une question délicate rencontrée en pharmacologie, à savoir la chiralité des acides aminés constitutifs des peptides thérapeutiques, qui représente une caractéristique structurelle subtile très difficile à sonder, en particulier à l’état de traces (0,1 %). Le projet GLYMS porte sur le développement de méthodes d’analyses qualitatives et quantitatives des peptides présents dans des milieux biologiques très complexes.
En rassemblant l’expertise nécessaire dans les technologies séparatives, la spectrométrie de masse, et en possédant les échantillons appropriés (bibliothèque de peptides faite maison) ou en étant capable de préparer toutes les séquences requises, le projet PRME GLYMS vise à se concentrer sur la distinction ultime et très subtile des séquences diastéréo-isomériques (mêmes compositions élémentaires et structures primaires) pour contrôler l’intégrité chirale des peptides thérapeutiques. Deux acides aminés connus pour leur tendance à l’épimérisation (l’acide aspartique et l’histidine) seront sélectionnés pour des études modèles. La remise en question de l’activité optique sur la caractérisation des peptides implique que la glycine, le seul résidu non chiral, doit également être incluse pour permettre une vue d’ensemble complète de toutes les caractéristiques critiques.
Remodelage enzymatique de la matrice extracellulaire
Remodelage enzymatique de la matrice extracellulaire : Expériences en milieux synthétiques encombrés et modèles cinétiques – X-CROWD (AAPG 2021 ANR – 2021 2025). Partenaire
La matrice extracellulaire (MEC) est essentielle pour le fonctionnement des organismes multicellulaires. Des maladies majeures, comme le cancer, sont associées à l’altération de son remodelage dynamique régulé par voie enzymatique. Cependant, celui-ci est encore peu compris au niveau théorique dans des conditions réalistes. Dans ce projet, nous étudierons comment la cinétique de certaines enzymes de la MEC est influencée par des milieux artificiels contrôlés incluant des composants clés de la MEC, qui sont aussi des substrats pour les enzymes, comme le collagène et l’acide hyaluronique. En partant de travaux collaboratifs préliminaires, nous réaliserons des analyses par spectroscopie UV/Vis, électrophorèse capillaire couplée à la mobilité ionique et à la spectrométrie de masse et RMN PFG sur des systèmes de complexité croissante. À terme, ce projet produira d’innovants modèles cinétiques de dégradation/remodelage enzymatique de composants clés de la MEC en conditions proches du réel.
Implication de l’équipe : Étude des changements de conformation de l’hyaluronidase, de l’élastase et de la collagénase induits par des milieux encombrés.
Les changements de conformation sont étudiés par infusion directe IMS-MS dans différents environnements : physiologique, dénaturant et en présence d’agents d’encombrement artificiels (Dextran PEG, PVP). Des données (distribution des états de charge et temps de dérive) relatives aux différentes conformations (extrêmes) des enzymes sont acquises et constituent une première échelle de paramètres IMS-MS. Ensuite, l’électrophorèse capillaire avec ionisation par électronébulisation (CESI) est couplée à l’IMS-MS pour étudier les changements de conformation des enzymes dans des environnements artificiellement et endogène encombrés en l’absence et en présence de leurs substrats. Le capillaire est d’abord utilisé comme un nanoréacteur enzymatique. Les différents composants sont séparés par électrophorèse et identifiés par SM. La distribution des états de charge acquise et le temps de dérive des enzymes par rapport à l’échelle précédemment établie ainsi que les mesures de mobilité électrophorétique permettent d’extraire des informations précieuses sur les changements de conformation induits par les différents milieux considérés.
Développement d’outils analytiques miniaturisés pour le Suivi Thérapeutique de Médicaments
Le Suivi Thérapeutique des Médicaments (Therapeutic Drug Monitoring, TDM) est une pratique clinique consistant au suivi des concentrations en substance active à des intervalles déterminés pour maintenir une concentration constante dans la circulation sanguine d’un patient en vue d’optimiser les schémas posologiques individuels. Cette démarche est fondée sur l’existence d’une relation entre la concentration en substance active et l’effet biologique. Cette pratique est particulièrement indiquée en cas d’administration de médicaments à index thérapeutique étroits ce qui correspond à des médicaments pour lesquels la différence entre la concentration active (responsable de l’efficacité thérapeutique) et la concentration toxique (responsable de l’apparition d’effets indésirables) est faible. Le TDM permet l’individualisation de la posologie du médicament en maintenant les concentrations plasmatiques ou sanguines du médicament dans une fenêtre thérapeutique ciblée. La mise en place d’un TDM permet une réduction des effets indésirables pour le patient ce qui a pour conséquence une diminution du coût de sa prise en charge pour les institutions.
Le TDM constitue un enjeu majeur de santé publique notamment pour le traitement des cancers. Ces dosages requièrent à ce jour le plus souvent l’utilisation d’équipements analytiques lourds et couteux (tels que la chromatographie liquide couplée à la spectroscopie UV ou la spectrométrie de masse en tandem (LC-MS/MS)), et la durée requise pour le rendu des résultats est longue (plusieurs jours entre la collecte des échantillons et le résultat), ce qui oblige le patient à se rendre régulièrement dans les services de santé qui sont saturés. Tous ces aspects rendent difficiles la mise en place d’un TDM mettant ainsi les patients en danger.
Le projet en cours vise à développer un nouvel outil analytique simple, rapide, peu couteux et autonome permettant la mesure directe par le patient des concentrations sanguines de différentes molécules thérapeutiques en vue d’individualiser en temps réel les adaptations posologiques. Le travail de thèse aura pour objectif le développement des systèmes analytiques miniaturisés (basés sur la technologie µfluidique) dédiés à l’analyse de concentrations plasmatiques de médicaments nécessitant un TDM personnalisé. Les systèmes développés devront répondre à l’un des principaux enjeux du TDM, à savoir un fonctionnement totalement intégré, de la préparation d’échantillon à la détermination de la concentration plasmatique.
Un dispositif microfluidique fabriqué à partir de la technologie laser et utilisant une résine innovante aux propriétés physico-chimiques particulières, sera développé. La miniaturisation de la préparation d’échantillon (précipitation des protéines, dessalage, extraction) sera réalisée pour la première fois (jamais publiée mais essais de faisabilité réalisés en interne) en combinant la technologie «Droplet microfluidics» et l’approche SALLE (Salting-out Assisted Liquid-Liquid Extraction).
Développement de méthodes analytiques pour l’étude de conjugués protéiques innovants entre des VHH et des oligonucléotides
Développement de méthodes analytiques pour l’étude de conjugués protéiques innovants entre des VHH et des oligonucléotides : caractérisation, stabilité et quantification dans des milieux biologiques. Partenariat industriel
Vect-Horus est une société privée de biotechnologie qui conçoit et développe des vecteurs facilitant l’administration ciblée de molécules thérapeutiques et d’agents d’imagerie. En particulier, Vect-Horus a développé et optimisé une famille de vecteur de type VHH capables de cibler le récepteur à la transferrine (TfR). Dans le cadre de ses activités de R&D, Vect-Horus conjugue ces VHHs à des oligonucléotides thérapeutiques afin d’en améliorer le potentiel par une augmentation de leur délivrance dans les tissus cibles surexprimant le TfR. Différents designs de conjugués ont été testés et ont permis de mettre en évidence un format hétérodimérique utilisant le fragment Fc comme plateforme. Le design retenu est basé sur une région Fc de type « nœud dans la cavité » fusionnée d’une part à un VHH ciblant le TfR et de l’autre à une étiquette peptidique utilisée pour la conjugaison par chimie enzymatique à de petits ARNs interférants (siRNA). Ces conjugués VHH-hFc-siRNA ont montré leur capacité d’extinction du gène cible dans les tissus d’intérêts. Vect-Horus souhaite maintenant développer les outils analytiques de caractérisation et quantification de ces conjugués afin d’en étudier la stabilité chimique et biologique, de connaitre le profil de dégradation de ces conjugués et d’être en mesure de quantifier la molécule intacte et ses métabolites dans différents milieux biologiques tels que le tritosome ou les tissus d’intérêt.
Ce projet de thèse va donc être constitué de plusieurs volets : dans un 1er temps, la bibliographie existante sur les techniques analytiques disponibles des conjugués de VHHs d’une part et d’oligonucléotides d’autre part, sur leur stabilité in vitro et in vivo et les méthodes appliquées va être recherchée. L’objectif sera de prendre appui sur les techniques existantes pour développer celles qui seront les plus adaptées à nos conjugués originaux.
Dans un second temps, sera développée une méthode stable et robuste pour la caractérisation du conjugué VHH-hFc-siRNA dans son tampon de conservation. Cela permettra d’obtenir le profil d’impureté du conjugué selon le processus de fabrication interne et constituera la base pour la suite.
Ensuite l’étude portera sur la dégradation du conjugué VHH-hFc-siRNA dans différentes conditions (conditions de stockage et conditions forcées). Le suivi de la dégradation du conjugué sera fait sur chaque condition durant 18 mois ainsi que les produits de dégradation potentiels.
Avec les informations obtenues par l’étude de stabilité en milieu tamponné, le suivi de la stabilité en milieu biologique (plasma/sang/urine) artificiellement dopé avec le conjugué sera réalisé et si besoin la méthode sera optimisée pour augmenter la sensibilité vis à vis du composé. La préparation de l’échantillon, sa détection et sa quantification à l’aide d’un étalon interne permettront de valider la méthode.
Enfin, la méthode validée sera utilisée dans le cadre d’une étude pharmacocinétique du conjugué VHH-hFc-siRNA chez la souris ainsi que la mise au point de la quantification du conjugué dans les tissus d’intérêt afin d’évaluer sa biodistribution in vivo.
Ces données seront cruciales pour anticiper le développement du conjugué VHH-hFc-siRNA par la société Vect-Horus. Cela ouvrira en outre la possibilité d’étudier la biodistribution de ce type de conjugué et ainsi offrira la possibilité d’utiliser cette méthodologie pour l’optimisation des conjugués VHH-hFc-siRNA dans différentes applications.
Chémobiologie des Venins et Toxines
Les plantes, les microbes et les animaux ont largement exploité ce monde invisible de « la chémobiologie » pour faciliter leur survie. Parmi les exemples les plus frappants de telles interactions chimiques ciblées, les animaux venimeux ont développé des molécules remarquables (« toxines ») au cours de leur évolution afin de faciliter la capture de leur proie et pour se défendre contre les prédateurs. Ces toxines, souvent de nature peptidique et riches en ponts disulfures, montrent des propriétés exceptionnelles en termes de stabilité, affinité et sélectivité pour leurs cibles. Ces dernières étant majoritairement des protéines membranaires ou des enzymes clé de notre physiologie, certaines toxines se sont révélées de précieux outils pharmacologiques pour les étudier, mais elles possèdent parfois également un fort potentiel de valorisation.
Bien que les venins animaux représentent une source très prometteuse pour la découverte de nouveaux composés d’intérêt, démêler leur complexité et isoler les composants actifs individuels du mélange constituant chaque venin est vrai challenge. En effet, un venin comprend des centaines de toxines peptidiques, ainsi que de nombreuses protéines, acides aminés et autres petites molécules. Avec plus de 200 000 espèces animales venimeuses, les venins s’apparentent une bibliothèque naturelle de millions de composés bioactifs. On estime que, selon les groupes considérés (serpents, scorpions, araignées, cônes…), seulement 0.05% à 2% de la diversité a été exploré, ouvrant un vaste champ d’investigation.
Ainsi, les thématiques de recherche développées depuis plus de 10 ans au CNRS, s’articulent autour de 3 axes principaux :
i) l’accélération de la découverte de nouvelles biomolécules issues des venins animaux en utilisant les technologies « omics », ii) leur synthèse, repliement et optimisation par voie chimique, et enfin, iii) la caractérisation de leur activité biologique afin de déterminer leur potentiel comme outil pharmacologique, mais également thérapeutique, agrochimique ou cosmétique.
Dans une vision plus globale, j’essaie également de replacer ces éléments dans un contexte qui évolue rapidement, notamment avec l’arrivée à maturité de certains outils de prédictions guidés par l’IA qui permettent d’obtenir des structures tridimensionnelles de protéines à partir de séquences primaires. En effet, la problématique majeure de ces projets de recherche reste la synthèse de l’isomère conformationnel natif et biologiquement actif. Ainsi, la prédiction de l’appariement des ponts disulfures, et plus encore l’ordre dans lequel ceux-ci doivent être réalisés devra permettre de nouvelles avancées dans le domaine.


 Christine ENJALBAL
Christine ENJALBAL










