Accueil du site > Recherche > PeptiSystems
Projet ANR PeptiSystems (2014–2019)
Processus énergétiques gouvernant des protométabolismes peptidiques à l’origine des systèmes vivants
PeptiSystems est un projet de recherche collaboratif sur la chimie prébiotique et les origines de la vie entre les équipes DSBC (Montpellier) et PIIM (Marseille), financé par l'Agence Nationale de la Recherche (ANR) sur quatre ans, projet n° ANR-14-CE33-0020.
Contours du projet
La détection d’exoplanètes en nombre grandissant et présentant de plus en plus des conditions voisines de celles de la Terre place la question de la pluralité des mondes vivants au tout premier plan des questions non résolues par la science. Cependant, tant l’étude de la nature et de l’émergence possibles d’autres formes de vie que leur détection réclament une approche interdisciplinaire. La vie terrestre constitue un processus historique qui s’est développé par complexification à partir de systèmes chimiques. Le projet PeptiSystems adopte l’idée que tant des forces motrices physico-chimiques que la contingence sont à l’origine de ce processus dès son début.
Une force motrice vers la complexité
Les aspects théoriques constitueront à la fois le point de départ du projet et un thème d’investigation destiné à mieux comprendre l’origine physique de la complexité. Dans cette perspective, l’émergence de protométabolismes définis comme des réseaux de réactions chimiques fonctionnant hors équilibre et donc capables d’induire une auto-organisation (associée à une diminution locale d’entropie compensée par l’irréversibilité du processus global) est une étape clef pour l’origine de la vie.
Réseaux de réactions irréversibles
Des réseaux chimiques de ce type doivent fonctionner à sens unique (de manière cinétiquement irréversible) et être constitués de suites de réactions consécutives ou de cycles réactionnels et peuvent impliquer en sus d’autres rétroactions positives donnant naissance à des systèmes doués de propriétés auto-catalytiques et se comportant de manière non-linéaire. Pour comprendre comment l’auto-organisation peut s’établir spontanément, il est essentiel de déterminer les voies ayant pu alimenter de tels systèmes en énergie de manière constante ou répétée.
Les biopolymères
Cette approche est appliquée à la formation de biopolymères potentiellement capables d’activités fonctionnelles qui est considérée le plus souvent comme un préalable à l’origine de la vie. Notre objectif original est de proposer un scénario global intégrant la formation de peptides et d’autres processus associés pour construire un ensemble donnant naissance à des propriétés émergentes liées à l’interconnexion des différentes parties du réseau réactionnel.
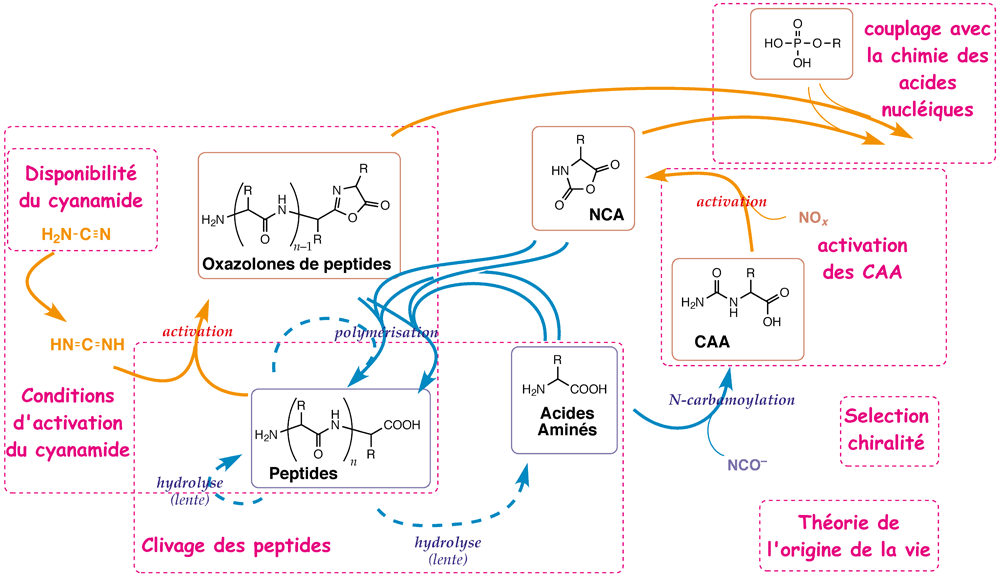
Le réseau de réactions chimiques formant un protométabolisme des peptides
qui sera étudié en détail dans le cadre du projet
Formation des peptides et traduction
Le projet PeptiSystems cible (i) la compréhension de la formation abiotique de peptides et le couplage de sources d’énergie à la formation de la liaison peptidique. Mais il n’est pas limité à ce but car des peptides obtenus à partir de mélanges racémiques ne sont généralement pas capables d’adopter les structures bien définies nécessaires à des activités spécifiques. Il s’intéressera (ii) aux questions de l’émergence de la sélectivité (stéréosélectivité et séquences de structure ou de chiralité définie) et d’états improbables mais cinétiquement stables.
Enfin (iii) l’émergence de la traduction à un stade précoce de l’évolution suggère que les chimies des acides aminés et des peptides peuvent être couplées à celle d’un support d’information en conformité avec l’hypothèse d’une co-évolution peptides – nucléotides. L’amélioration des connaissances des voies chimiques par lesquelles des acides aminés ou des peptides courts ont pu interagir avec des ribonucléotides (monomères ou oligomères), est nécessaire pour comprendre l’émergence des processus de traduction, et constitue une priorité dans nos travaux.
L’ensemble de ces thèmes font l’objet d’une étude expérimentale basée sur le suivi des réactions par des méthodes classiques de chimie organique et analytique (HPLC, spectrométries RMN, masse, UV…).
Personnes impliquées dans le contrat ANR :
Équipe DSBC de l’IBMM (Montpellier)
Permanents :
Robert Pascal (Directeur de Recherche émérite CNRS, coordinateur du projet) [rattaché à PIIM/ASTRO à/c du 2019-05-01]
Laurent Boiteau (Chargé de Recherche CNRS)
Jean-Christophe Rossi (Maître de Conférences, Université de Montpellier)
Anciens :
Damien Beaufils (doctorant MESR, thèse soutenue le 06 nov. 2015)
Ziwei Liu (Simons Foundation postdoctoral fellow, nov. 2013 – jan 2017)
Ghinwa Ajram (doctorante ANR, thèse soutenue le 29 nov. 2018)
Équipe ASTRO du laboratoire PIIM (Marseille) [site web]
Permanents :
Grégoire Danger (Professeur, Université d’Aix-Marseille, membre junior honoraire IUF)
Fabrice Duvernay (Maître de Conférences, Université d’Aix-Marseille)
Collaborations externes (hors financement ANR)
Laboratoire SQPOV, UMR 408 INRA, UAPV Université d’Avignon et des Pays de Vaucluse, France
– Dr. Raphaël Plasson
Ben Gurion University of Negev, Israel
– Prof. Addy Pross [site web]
MRC Laboratory of Molecular Biology, Cambridge, UK
[site web]
– Prof. John D. Sutherland (group leader),
– Dr. Ziwei Liu (postdoctoral fellow, depuis mars 2017),
– Dr. Dougal Ritson (career development fellow)
IAS (Information and Autonomous Systems) Research Centre for Life, Mind & Society,
EHU-UPV Université du Pays Basque, Espagne
[site web]
– Prof. Kepa Ruiz-Mirazo (group leader),
– Sara Murillo Sánchez (doctorante, thèse soutenue le 07 sept. 2017)
UCSB University of Califdornia Santa Barbara & NASA, USA
– Prof. Irene A. Chen
Laboratoire ICBMS, UMR 5246 CNRS, Université Claude-Bernard Lyon 1, France
– Prof. Pierre Strazewski (group leader),
– Dr. Michele Fiore (Maître de Conférences)
Publications
(les articles “Open Acess” sont repérés par une icône sur le lien DOI)
R. Pascal,
Kinetic barriers and self-organisation of life.
Isr. J. Chem. 2015, 55, 865–874.
DOI: 10.1002/ijch.201400193
R. Pascal, A. Pross,
Stability and its manifestation in the chemical and biological worlds.
Chem. Commun. 2015, 51(90), 16160–16165.
DOI: 10.1039/C5CC06260H
D. Beaufils, S. Jepaul, Z. Liu, L. Boiteau, R. Pascal,
The activation of free dipeptides promoted by strong activating agents in water does not yield diketopiperazines.
Orig. Life Evol. Biosph. 2016, 46(1), 19–30.
DOI: 10.1007/s11084-015-9455-0
S. Murillo Sánchez, D. Beaufils, J. M. González Mañas, R. Pascal, K. Ruiz-Mirazo,
Fatty acids’ double role in the prebiotic formation of a hydrophobic dipeptide.
Chem. Sci. 2016, 7(5), 3406–3413.
DOI: 10.1039/c5sc04796j
R. Pascal,
Physicochemical requirements inferred for chemical self-organization hardly support an emergence of life in the deep ocean of icy moons.
Astrobiology 2016, 16, 328–334.
DOI: 10.1089/ast.2015.1412
 Z. Liu, L. Rigger, J.-C. Rossi, J. D. Sutherland, R. Pascal,
Z. Liu, L. Rigger, J.-C. Rossi, J. D. Sutherland, R. Pascal,
Mixed anhydride intermediates in the reaction of 5(4H)-oxazolones with phosphate esters and nucleotides.
Chem. Eur. J. 2016, 22(42), 14940–14949.
DOI: 10.1002/chem.201602697
□ 2e de couverture (inside cover).
Chem. Eur. J. 2016, 22(42), 14758.
DOI: 10.1002/chem.201603523
R. Pascal, A. Pross,
The logic of life.
Orig. Life Evol. Biosph. 2016, 46(4), 507–513.
DOI: 10.1007/s11084-016-9494-1
R. Pascal, A. Pross,
A roadmap toward synthetic protolife.
Synlett 2017, 28(01), 30–35.
DOI: 10.1055/s-0036-1589403
Z. Liu, C. Hanson, G. Ajram, L. Boiteau, J.-C. Rossi, G. Danger, R. Pascal,
5(4H)-Oxazolones as effective aminoacylation reagents for the 3’-terminus of RNA.
Synlett 2017, 28(01), 73–77.
DOI: 10.1055/s-0036-1588647
A. Pross, R. Pascal,
How and why kinetics, thermodynamics, and chemistry induce the logic of biological evolution.
Beilstein J. Org. Chem. 2017, 13, 665–674.
DOI: 10.3762/bjoc.13.66
N. Abou Mrad, G. Ajram, J.-C. Rossi, L. Boiteau, F. Duvernay, R. Pascal, G. Danger,
The prebiotic C-terminal elongation of peptides can be initiated by N-carbamoyl amino acids.
Chem. Eur. J. 2017, 23(31), 7418–7421.
DOI: 10.1002/chem.201700702
Z. Liu, J.-C. Rossi, R. Pascal,
How prebiotic chemistry and early life chose phosphate.
Life 2019, 9(1), 26.
DOI: 10.3390/life9010026
Z. Liu, G. Ajram, J.-C. Rossi, R. Pascal,
The chemical likelihood of ribonucleotide-α-amino acid copolymers as players for early stages of evolution.
J. Mol. Evol. 2019, 87(2–3), 83–92.
DOI: 10.1007/s00239-019-9887-7
A. D. Pressman, Z. Liu, E. Janzen, C. Blanco, U. F. Müller, G. F. Joyce, R. Pascal, I. A. Chen,
Mapping a systematic ribozyme fitness landscape reveals a frustrated evolutionary network for self-aminoacylating RNA.
J. Am. Chem. Soc. 2019, 141(15), 6213–6223.
DOI: 10.1021/jacs.8b13298
R. Pascal,
A possible prebiotic basis for metabolism.
[invited News & Views commentary]
Nature 2019, 569, 47–49.
DOI: 10.1038/d41586-019-01322-3
R. Pascal, A. Pross,
Chemistry’s kinetic dimension and the physical basis for life.
J. Syst. Chem. 2019, 7, 1–8.
DOI n/c
G. Ajram, J.-C. Rossi, L. Boiteau, R. Pascal,
A reassessment of prebiotically relevant chemical agents for the activation of α-amino acids and peptides.
J. Syst. Chem. 2019, 7, 19–28.
DOI n/c
R. Pascal, I. A. Chen,
From soup to peptides.
[invited News & Views commentary]
Nat. Chem. 2019, 11, 763–764.
DOI: 10.1038/s41557-019-0318-6
liens
- Agence Nationale de la Recherche
- Physique des Interactions Ioniques et Moléculaires – UMR7345, Marseille
- Société Française d’Exobiologie
- ISSOL – The International Astrobiology Society
- EU COST Chemistry Action CM1304 Emergence and Evolution of Complex Chemical Systems
- EU COST TransDomain Action TD1308 Life-Origins
- Simons Collaboration on the Origins of Life (Simons Foundation, USA)

L’équipe PeptiSystems de Montpellier en juin 2014
(de gauche à droite : Jean-Christophe, Sara, Laurent, Damien, Robert, Ziwei)